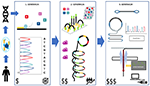
Od Sangera do sekwencjonowania genomów – przegląd technologii sekwencjonowania DNA
DOI:
https://doi.org/10.18388/pb.2021_534Abstrakt
Nie ma techniki, która wniosłaby większy wkład w rozwój genetyki, biologii molekularnej i medycyny niż sekwencjonowanie DNA. Przez wiele lat złoty standard w tym zakresie stanowiła metoda oparta na enzymatycznej syntezie DNA opracowana przez Frederica Sangera. Pod koniec XX. wieku nastąpił dynamiczny rozwój technologii sekwencjonowania nowej generacji (NGS), które zakończyły erę analizy pojedynczych genów i zapoczątkowały erę sekwencjonowania genomów. Pomimo ostrej konkurencji, jedna z technologii NGS praktycznie całkowicie zdominowała światowy rynek. W artykule przedstawiamy autorski przegląd metod sekwencjonowania DNA, począwszy od metody Sangera po wysokoprzepustowe technologie sekwencjonowania drugiej i trzeciej generacji, ze szczególnym uwzględnieniem tych, które odniosły komercyjny sukces. Prezentujemy ich krótką historię, zasady działania, możliwości techniczne, zastosowania i ograniczenia. W podsumowaniu zdradzamy ile kosztuje sekwencjonowanie genomu człowieka na obecnym etapie genomicznej rewolucji i nakreślamy perspektywy dalszego rozwoju genomiki.
Pobrania
Opublikowane
Licencja
Prawa autorskie (c) 2024 Małgorzata Marcinkowska-Swojak, Magdalena Rakoczy, Jan Podkowiński, Jurand Handschuh, Paweł Wojciechowski, Luiza Handschuh

Utwór dostępny jest na licencji Creative Commons Uznanie autorstwa 4.0 Międzynarodowe.
Zawartość kwartalnika jest rozpowszechniana na licencji Creative Commons Uznanie autorstwa 4.0 Międzynarodowe (CC BY 4.0). Uznanie autorstwa — Utwór należy odpowiednio oznaczyć, podać link do licencji i wskazać jeśli zostały dokonane w nim zmiany . Możesz to zrobić w dowolny, rozsądny sposób, o ile nie sugeruje to udzielania przez licencjodawcę poparcia dla Ciebie lub sposobu, w jaki wykorzystujesz ten utwór.
Prawa autorskie do prac © pozostają przy autorach.
Prawa autorskie do czasopisma © posiada Polskie Towarzystwo Biochemiczne.