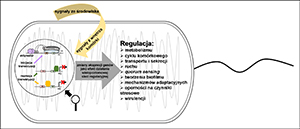
Rola regulatorów transkrypcji w kontroli ekspresji genów prokariotycznych na przykładzie modelowej bakterii Pseudomonas aeruginosa
DOI:
https://doi.org/10.18388/pb.2021_490Abstrakt
Bakterie Pseudomonas aeruginosa spotykane są często w środowisku naturalnym jak woda czy gleba, ale zaliczane są także do oportunistycznych patogenów ludzi i zwierząt. Charakteryzuje je duża zdolność do przetrwania w bardzo różnych niszach ekologicznych. Adaptacja i umiejętność dostosowania do zmiennych warunków środowiska wynika z rozbudowanych sieci regulacyjnych tych bakterii i wykorzystania bogatego repertuaru kodowanych w genomie białek, ścieżek metabolicznych, mechanizmów obronnych i adaptacyjnych.
Regulatory transkrypcji są kluczowymi elementami regulacji ekspresji genów, które odpowiadają na sygnały z otoczenia, włączając lub wyłączając określone ścieżki. Kompleksowe badania regulatorów transkrypcji z wykorzystaniem metod transkryptomicznych, genomicznych, regulacyjnych dostarczają wiedzy na temat mechanizmów ich działania, regulowanych genów i procesów, umożliwiając poznanie i zrozumienie skomplikowanych często sieci regulacyjnych zawiadujących przeżyciem komórki w danym środowisku i warunkach.
Celem niniejszej pracy jest przedstawienie kluczowych zagadnień związanych z regulacją transkrypcji bakteryjnej na podstawie przeglądu danych literaturowych oraz zobrazowanych na przykładzie P. aeruginosa i charakterystyki mechanizmów regulacji ekspresji genów zaangażowanych między innymi w wirulencję tej bakterii.
Pobrania
Opublikowane
Numer
Dział
Licencja
Prawa autorskie (c) 2023 Magdalena Modrzejewska-Balcerek, Aneta Agnieszka Bartosik

Utwór dostępny jest na licencji Creative Commons Uznanie autorstwa 4.0 Międzynarodowe.
Zawartość kwartalnika jest rozpowszechniana na licencji Creative Commons Uznanie autorstwa 4.0 Międzynarodowe (CC BY 4.0). Uznanie autorstwa — Utwór należy odpowiednio oznaczyć, podać link do licencji i wskazać jeśli zostały dokonane w nim zmiany . Możesz to zrobić w dowolny, rozsądny sposób, o ile nie sugeruje to udzielania przez licencjodawcę poparcia dla Ciebie lub sposobu, w jaki wykorzystujesz ten utwór.
Prawa autorskie do prac © pozostają przy autorach.
Prawa autorskie do czasopisma © posiada Polskie Towarzystwo Biochemiczne.